1. クライオ電顕像からのタンパク質構造モデリング
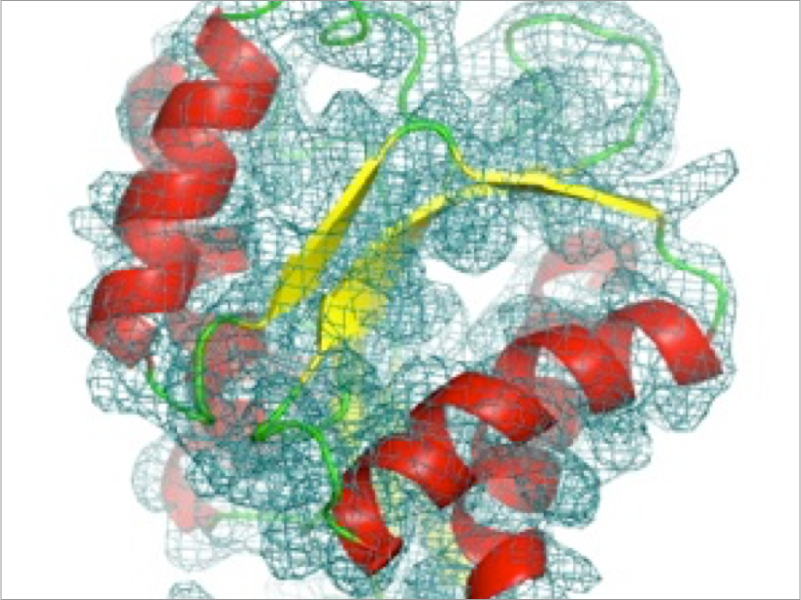
近年、クライオ電子顕微鏡を用いた単粒子解析法によるタンパク質の立体構造解析が盛んに行われています。タンパク質の結晶化を必要としないため、これまでX線結晶構造解析で解くことが難しかった巨大タンパク質複合体の立体構造が、近原子解像度で続々と決定されています。単粒子解析法により得られたタンパク質複合体の立体像から、分子構造をモデリングするためには、X線結晶構造解析やNMRなどで決定されたタンパク質の分子構造を、電子顕微鏡像にドッキングまたはフィッティングする方法が有効です。私たちは巨大生体分子複合体の精密な構造モデリングを確立することを目指し、MD計算プログラムGENESISを基盤としたフィッティング法の開発と応用を行っています。
2. データ科学に基づくタンパク質ダイナミクスの理解
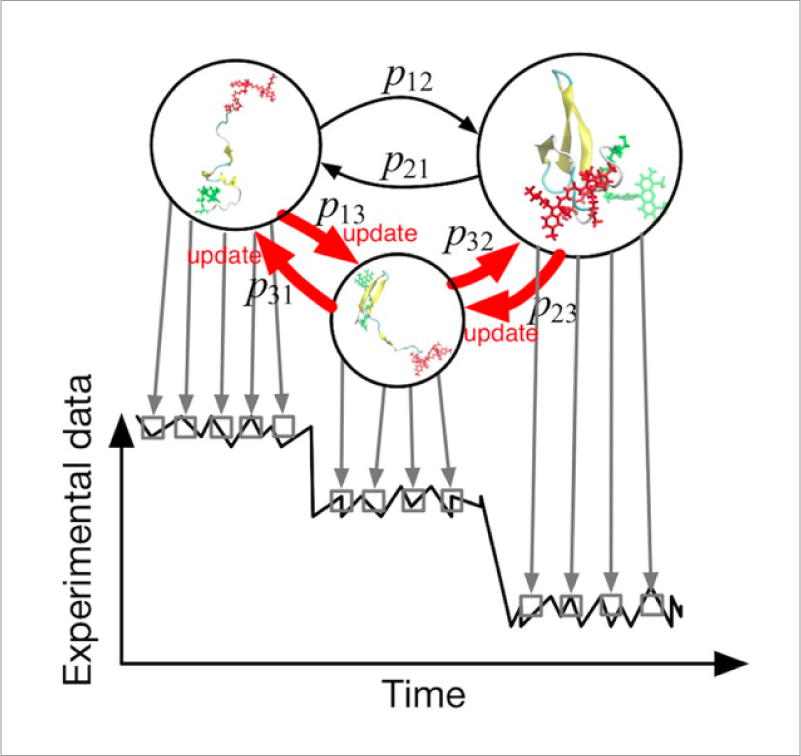
タンパク質構造の経時変化は、酵素反応やシグナル伝達などの細胞機能に深く関わっています。これらを理解するためには、蛋白質の構造ダイナミクスを高解像度で解析する手法が必要です。MD計算を用いれば原子レベルでの情報を得ることができますが、モデルのパラメータは完全に正しいとは限らず、その不正確さがダイナミクスに現れることもあります。一方、1分子FRETを用いれば構造揺らぎを時系列データとして観測できますが、距離情報のみから分子構造を解釈するのは困難です。解像度の粗い計測データと原子モデルのMD計算を相補的に組み合わせて実験データを解釈するために、私たちは機械学習やデータ同化などを用いた新しい手法を開発し、膜タンパク質などへの応用を行っています。
参考文献
- “Acceleration of cryo-EM Flexible Fitting for Large Biomolecular Systems by Efficient Space Partitioning”, Takaharu Mori, Marta Kulik, Osamu Miyashita, Jaewoon Jung, Florence Tama and Yuji Sugita, Structure, 27, 161-174 (2019).
- “Flexible Fitting to Cryo-EM Density Map Using Ensemble Molecular Dynamics Simulations”, Osamu Miyashita, Chigusa Kobayashi, Takaharu Mori, Yuji Sugita, and Florence Tama., J. Comp. Chem., 38, 1447-1461 (2017).
- “Linking time-series of single-molecule experiments with molecular dynamics simulations by machine learning”, Yasuhiro Matsunaga and Yuji Sugita, eLife, 7, e32668 (2018).
- “Sequential data assimilation for single-molecule FRET photon-counting data”, Yasuhiro Matsunaga, Akinori Kidera, and Yuji Sugita., J. Chem. Phys., 142, 214115 (2015).
- “Characterization of Conformational Ensembles of Protonated N-glycans in the Gas-Phase”, Suyong Re, Shigehisa Watabe, Wataru Nishima, Eiro Muneyuki, Yoshiki Yamaguchi, Alexander D. MacKerell Jr. and Yuji Sugita, Scientific Reports, 8, 1644 (2018).
共同研究者
宮下治 (理研R-CCS)、Florence Tama (理研R-CCS, 名古屋大)、松永康佑 (埼玉大)、佐甲靖志 (理研CPR)